
Markierung von Proteinen
In den letzten Jahrzehnten ist die Fluoreszenzmikroskopie in vielen wissenschaftlichen Bereichen wie der Zellbiologie und Medizin zu einem unverzichtbaren Werkzeug geworden. Im Vergleich zur herkömmlichen Durchlichtmikroskopie bietet sie einen höheren Kontrast und eine höhere Empfindlichkeit. Die Verwendung von spektral unterschiedlichen Fluoreszenzmarkern ermöglicht die gleichzeitige Visualisierung verschiedener Ziele innerhalb derselben Probe. Hochauflösende Mikroskopie (z.B. STED, dSTORM) und FRET sind einige der beliebtesten Fluoreszenztechniken, die einzigartige Einblicke in zelluläre Prozesse, Verteilung und Strukturdynamik von Proteinen im unteren Nanometerbereich, weit unterhalb der natürlichen Beugungsgrenze des Lichts, ermöglichen.
Es gibt mehrere Ansätze, wie man ein Protein von Interesse (POI) fluoreszierend markiert, nämlich direkte Konjugation mit einem fluoreszierenden organischen Farbstoff, Fusion von POI mit selbstmarkierenden Tags (SLP) oder fluoreszierenden Proteinen und Immunfärbung. Für sie alle ist es entscheidend, eine Veränderung der Funktion des Proteins bei der Markierung zu verhindern. Während die Entdeckung und Implementierung von fluoreszierenden Proteinen die Bildgebung von lebenden Zellen revolutionierte, sind ihre schlechten photophysikalischen Eigenschaften (schnelles Ausbleichen/geringe Helligkeit) und ihre relativ große Größe immer noch limitierende Faktoren für viele Anwendungen. Im Gegensatz dazu bietet eine breite Palette von kleinen organischen Fluoreszenzfarbstoffen mit hervorragenden photophysikalischen Eigenschaften eine bessere Alternative zur Kennzeichnung. Die traditionelle Biokopplung mit Fluoreszenzfarbstoffen nutzt zugängliche, natürlich vorkommende reaktive Gruppen (Thiole oder Amine) von Proteinen und entsprechenden reaktiven Fluorophor-Derivaten (Maleimid oder NHS-Ester, Abb. 1a).
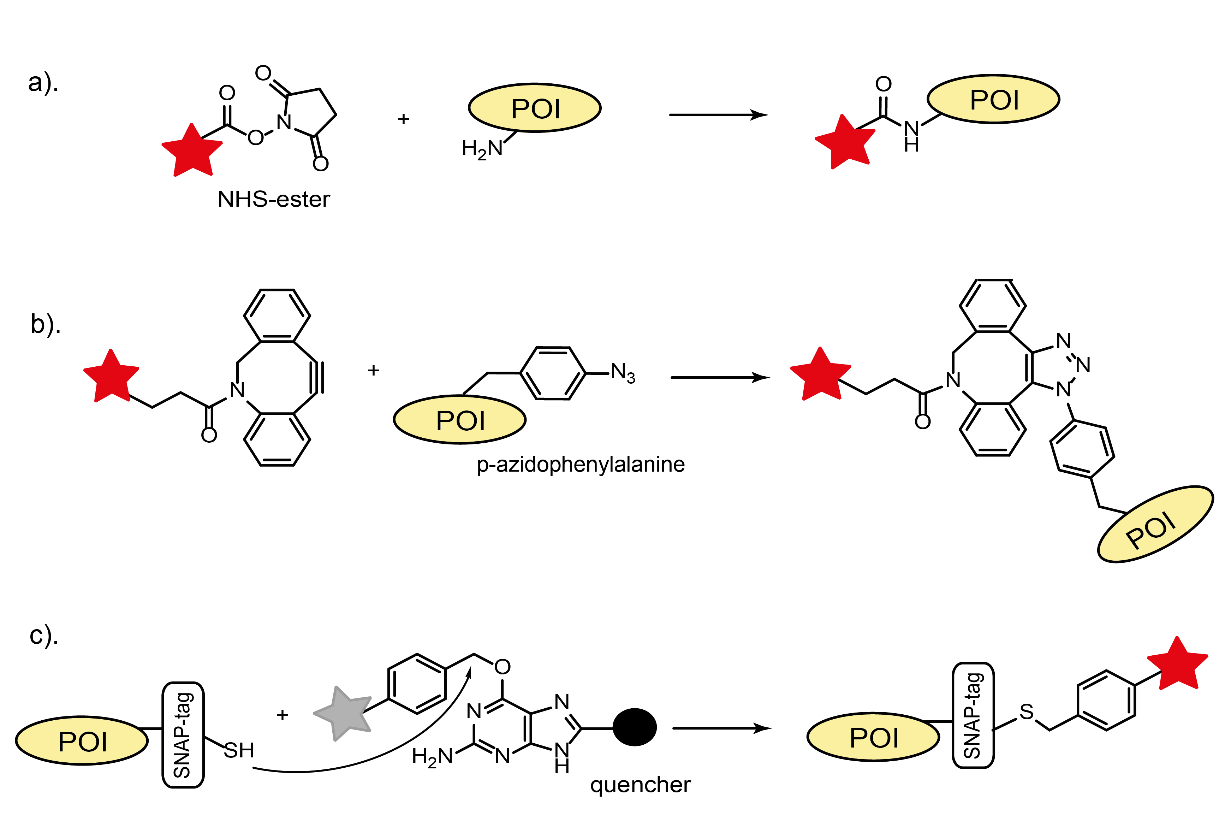
Diese direkte Konjugation von POIs mit Farbstoffen ist oft mit in vitro Untersuchungen und der Markierung von Antikörpern für das Immunostaining verbunden. Eine neu entwickelte bioorthogonale Kopplungsstrategie ermöglicht eine bessere Kontrolle der Standortspezifität und der Stöchiometrie. Sie basiert auf der Integration von unnatürlichen Aminosäuren, die einzigartige chemische Gruppen (z.B. para-Acetyl- oder para-Azideinheiten) tragen. Diese Gruppen reagieren sehr spezifisch mit ihren Gegenstücken in Oximkondensationen oder Click-Chemie-Reaktionen. Zusätzlich ermöglicht eine kupferfreie Klickreaktion in vivo Anwendungen (Abb. 1b). Die SLP-Technologie (Halo-, SNAP- und CLIP-Tags) vereint die Vorteile aller drei oben beschriebenen Markierungsmethoden. Es basiert auf der Bildung einer spezifischen kovalenten Bindung zwischen einem reaktiven Fluorophorderivat und einem SLP-Tag von fusionierten POIs (Abb. 1c). Diese Technik ist sowohl für lebende als auch für fixierte Zellen anwendbar. Die jüngste Entwicklung so genannter fluorogener Labels hat das Fluoreszenz-Toolkit noch weiter ausgebaut. Ein Markenzeichen dieser Marker ist ihre Fähigkeit, Fluoreszenz zu erzeugen, wenn sie an ein bestimmtes Ziel in vivo und in vitro gebunden sind. Dies führt zu einem höheren Bildkontrast durch den reduzierten unspezifischen Hintergrund. Die Fluorogenität kann durch Quenching-Effekte (PET oder FRET) der benachbarten Einheiten realisiert werden, die abgespalten werden oder bei der Markierungsreaktion eine strukturelle Veränderung erfahren (Abb. 1c). Die Kopplungschemie von unnatürlichen Aminosäuren oder selbstmarkierenden Tags kann für die fluorogene Proteinmarkierung genutzt werden. Je nach Anwendung kann eine Kombination aller dieser Kennzeichnungsmethoden eine Methode der Wahl sein.
Kontakt
GATTAquant GmbH
Staffelseestraße 8
DE-81477 München
Phone: +49 89 2153 720 80
INFO@GATTAQUANT.COM
Gefördert durch

Zahlungsmöglichkeiten


 Englisch
Englisch Deutsch
Deutsch Chinesisch
Chinesisch 